La bioinformatique est devenue aujourd’hui un domaine incontournable de la biologie. Ce champ de recherche multidisciplinaire est divisé en quatre catégories :
Pour manipuler, éditer ou analyser des jeux de séquences nucléiques ou protéiques, il existe de nombreux logiciels : certains généralistes et d’autres spécialisés pour répondre à des tâches précises (alignement de séquences, filtrage, etc.). Pour s’y retrouver dans cette masse de contenus, Biomanda vous propose de découvrir Seaview, un logiciel de manipulation de séquences généraliste, simple et efficace.
Seaview est un logiciel disponible sur Windows, Mac et Linux en version 32-bit et 64-bit. Il est développé par l’équipe du Dr. Manolo Gouy, au sein du Laboratoire de Biométrie et Biologie Evolutive (LBBE) situé à Lyon. Il peut être téléchargé sur le site du prabi à l’adresse suivante : http://doua.prabi.fr/software/seaview.
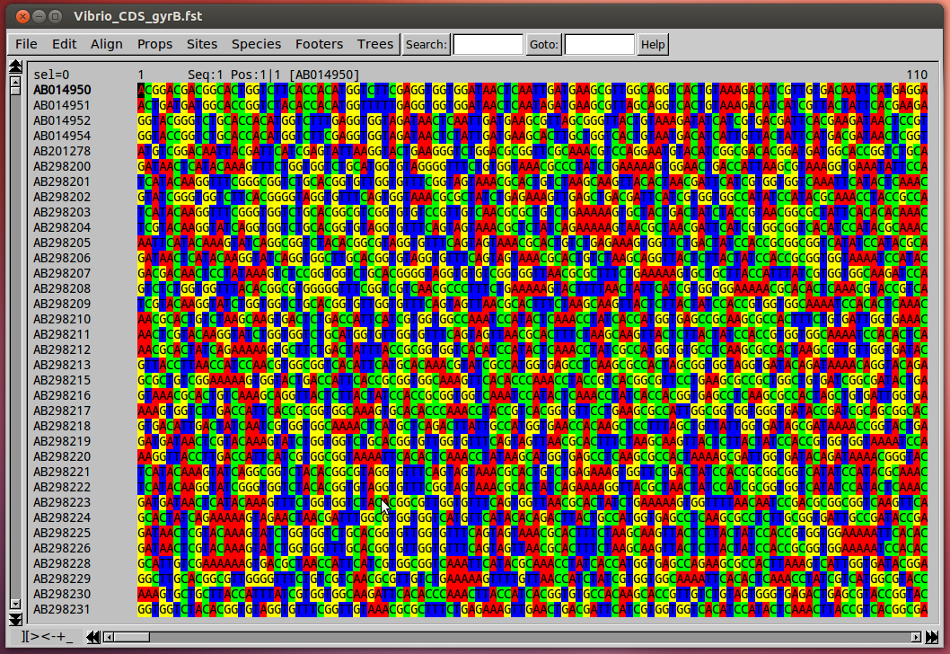
Seaview prend en charge de nombreux formats de fichiers comme Fasta, Clustal ou Nexus et permet de manipuler des séquences d’ADN, d’ARN ou de protéines. En fonction de la nature des séquences, un code couleur est automatiquement attribué à chaque lettre de manière à rapidement identifier les zones conservées et les zones variables des séquences. Le logiciel propose également tout un tas d’outils simples et pourtant extrêmement pratiques :
Il est également possible de dupliquer une séquence ou encore l’éditer en quelques clics. Pour ajouter des séquences supplémentaires, Seaview propose une fenêtre pop-up pour réaliser des copier-coller très facilement. Enfin l’option « View as protein » vous permettra en un clic de passer de séquences nucléiques à séquences protéiques.
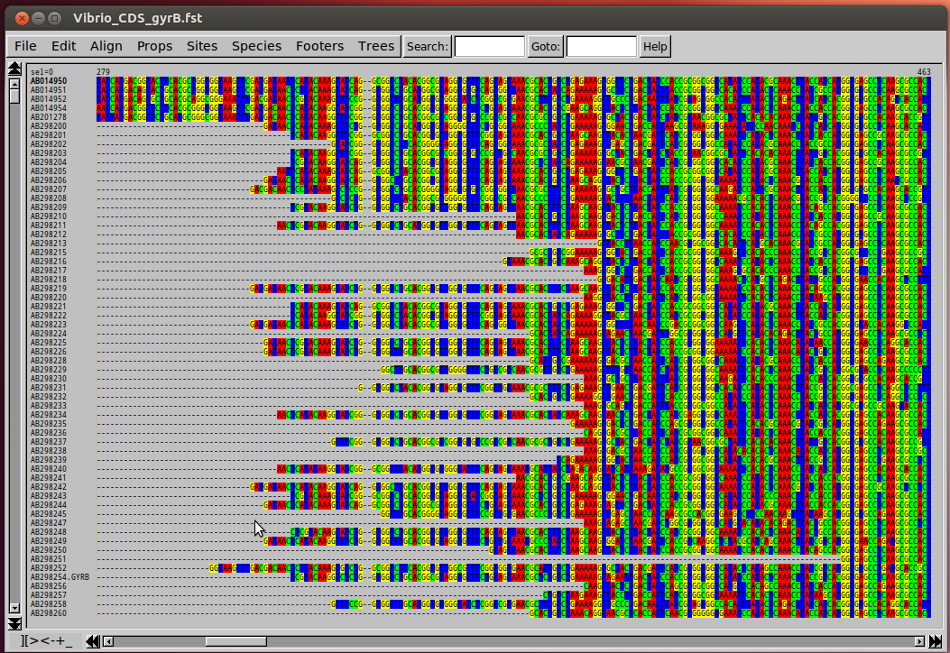
La base du projet Seaview est de proposer un environnement de travail pour manipuler les alignements de séquences. L’outil vous offre la possibilité d’aligner la totalité ou une partie de vos séquences, mais il est également possible de définir des zones dans les séquences qui pourront être alignées indépendamment. Cette fonction est très utile lorsque l’on souhaite par exemple corriger les erreurs liées aux algorithmes d’alignements de manière automatique. Enfin Seaview permet de facilement intégrer de nouvelles séquences à un alignement existant ou de fusionner plusieurs alignements de séquences en respectant le travail déjà réalisé sur chacun, grâce à la fonction d’alignement de profils. Mais l’atout du logiciel reste sa flexibilité en proposant d’utiliser Clustal et Muscle, ou d’ajouter d’autres algorithmes d’alignements. En effet, en fonction du jeu de données, du temps alloué et de la problématique biologique, le choix du programme est primordial pour obtenir une bonne qualité de résultat.
Générer de la donnée s’est bien mais l’organiser c’est mieux. Dans Seaview, vous pouvez créer des groupes de séquences, générer des sélections de zones génétiques et enfin annoter vos séquences. L’interface offre également de nombreux raccourcis pour déplacer des séquences d’un simple clic ou rajouter des INDELs dans une ou des séquence(s). Seaview utilise un format de fichier particulier, mase, qu’il faudra utiliser pour pouvoir conserver toutes ses modifications et sélections.
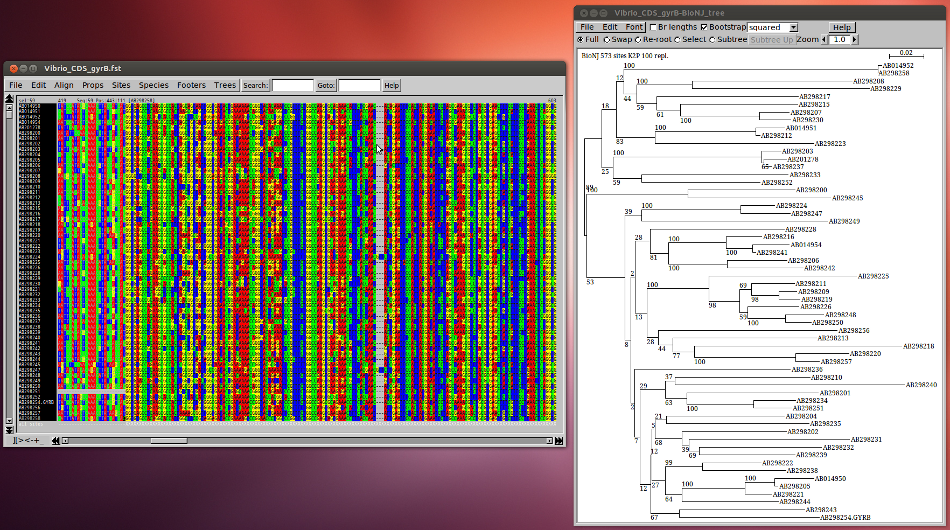
Depuis quelques années, Seaview a incorporé une interface d’analyses phylogénétiques qui permet de lancer une construction phylogénétique à partir d’une sélection de sites sur l’ensemble ou une partie des séquences. Il est possible d’utiliser la plupart des méthodes de reconstruction phylogénétique (parcimonie, distance, maximum de vraisemblance) à l’exception des inférences bayésiennes. Comme toujours, on retrouve flexibilité et personnalisation avec le menu de paramétrage des algorithmes de phylogénie qui permettra d’adapter ses besoins. L’application offre également l’opportunité de manipuler son arbre phylogénétique :
En outre, la fenêtre contenant votre arbre phylogénétique reste en communication avec le reste de l’interface et il est ainsi possible de réorganiser ses séquences en fonctions de la topologie de l’arbre, très utile lorsque l’on doit affiner un alignement ou définir des zones conservées. On regrettera de ne pas pouvoir plus annoter son arbre (changer le nom des feuilles, colorier, modifier la police, etc.), mais ces fonctionnalités devraient rapidement apparaître.